新冠疫苗的设计原理
在人类预防传染病的历史中,疫苗的发明具有里程碑意义,数百年来挽救了无数生命。新冠疫苗作用原理即通过人为引入病毒抗原,利用人体自身免疫系统,对靶抗原进行表位识别、免疫放大及记忆,进而产生持久且特异的体液及细胞免疫诱导出中和抗体清除入侵病毒的过程。根据靶抗原携带的表位序列,抗原呈递细胞将表位呈递给不同的T 细胞亚群,并将之进一步活化为辅助性T 细胞(Th 细胞)和细胞毒性T 细胞(Tc 细胞)。Th 细胞会协助B 细胞识别抗原、产生以抗体为代表的体液免疫,而Tc 细胞则能直接识别抗原,裂解被感染的宿主细胞,实现细胞免疫。
1.1基于SARS-CoV-2 致病机制的靶抗原选择
SARS-CoV-2内部的RNA 携带遗传信息,主导病毒的复制,而蛋白体由四部分组成,包括了S 蛋白(Spike protein)、E 蛋白(Envelope protein)、M 蛋白(Membrane protein)和N 蛋白(Nucleocapsid protein) 。其中,SARS-CoV-2 表面的S 蛋白能介导病毒入侵宿主细胞,机理是S 蛋白上的受体结合区(Receptor- binding domain,RBD)能识别宿主细胞上的血管紧张素转化酶2(Angiotensin-converting enzyme-2,ACE2) 。研究发现利用小分子抑制剂和病人体内的血清能够阻断S 蛋白与ACE2 的识别以及细胞入侵。而且,SARS-CoV-2 S 蛋白诱导的中和抗体能够显著阻断识别途径,降低病毒的侵染能力。这些研究充分说明了S 蛋白中和抗体对于预防病毒入侵的重要作用,为基于B 细胞体液免疫的疫苗设计提供了重要指导。与此同时,T 细胞免疫则对清除被感染细胞和病毒至关重要。因为一旦少数病毒逃脱了中和抗体的围捕,被感染的宿主细胞将成为病毒复制乃至变异的温床,而此时抗原特异性的Tc 细胞能够识别被侵染的宿主细胞,进一步清除病毒。研究发现,严重的COVID-19 病人体内出现了T 细胞耗竭以及功能多样性降低的现象,这反映了T 细胞免疫对于病情控制的重要性。
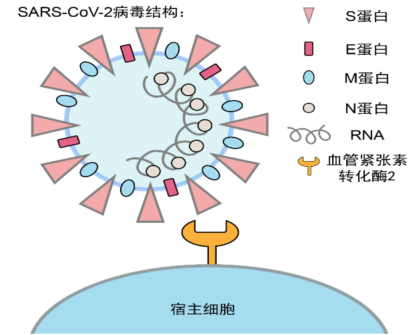
图1 SARS-CoV-2 结构组成及侵染细胞示意图
1.2靶蛋白的表位分析及其重要性
由于免疫反应的特异性,靶抗原表位序列(包括B 细胞表位和T 细胞表位)直接决定了免疫反应的种类。鉴于B 细胞免疫和T 细胞免疫都在SARS-CoV-2 的清除中不可或缺,尽可能去选择合适的免疫原性片段,是新冠疫苗设计的关键一步。由于含有多个B 细胞表位和T 细胞表位,全长的S 蛋白依然是目前新冠疫苗设计中最重要的靶抗原选择。然而,对SARS-CoV-2 与SARS-CoV 侵染后病人和小鼠的抗体种类进行评估后,人们发现S 蛋白中的保守表位会诱导出非中和抗体的产生。根据以往SARS-CoV 候选疫苗的研究,这类的抗体并不能起到有效的保护型免疫,反而有可能会加重抗体依赖感染增强效应(Antibody-dependent enhancement,ADE),引发病毒的扩大感染。值得注意的是,近期我国研究人员分析了COVID-19 病人体内的血清并发现:S 蛋白的RBD 片段含有多个B 细胞和T 细胞表位,并能够引发强烈的保护性抗病毒免疫;N 蛋白同样具有较高的免疫原性,是疫苗潜在靶点之一。这一发现为RBD 作为S 蛋白重要免疫原性区域提供有力支撑,为后续的亚单位疫苗设计提供思路。尽管全球疫苗研究正高速开展,但人们对SARS-CoV-2 的表位分析却报道较少。深入理解S 蛋白等蛋白靶标的表位分布及种类将为未来新冠疫苗设计提供重要理论基础。
1.3新冠疫苗的研发设计平台
疫苗设计步骤主要包括了靶抗原的合理选择、免疫增强佐剂的设计、体液免疫及细胞免疫类型的调控以及免疫流程的临床学优化。合适的药物研发平台是整合诸多设计步骤的重要基础。基于前期在SARS-CoV 以及MERS-CoV 疫苗上的安全性以及有效性验证,新冠疫苗拓展了新的平台技术,具体的开发策略包括了灭活及减活病毒疫苗、腺病毒载体疫苗、重组蛋白疫苗、核酸疫苗和其他疫苗(图2)。据报道,截至2021年4月2日,各国已向世界卫生组织报告的在研候选新冠疫苗超过200款,其中临床前研究184款,正在进行临床研究或批准使用的有85宽,我国已有5款疫苗已经批准紧急使用或附条件上市。

图2 新冠疫苗设计及研发平台示意图
参考文献:
[1]李文浩,李艳梅.大学化学[J]. 学化学[,35(12),29,29台
[2] Shang, J.; Ye, G.; Shi, K.; Wan, Y.;Luo, C.; Aihara, H.; Geng, Q.; Auerbach, A.; Li, F. Nature 2020, 581, 221.
[3] Hoffmann, M.;Kleine-Weber, H.; Schroeder, S.; Kruger, N.; Herrler, T.; Erichsen, S.; Schiergens, T. S.; Herrler, G.; Wu, N. H.; Nitsche, A.; et al.Cell 2020, 181, 271.
[4]Letko, M.; Marzi, A.; Munster, V. Nat. Microbiol. 2020, 5, 562.
[5]Rueckert, C.; Guzman, C. A. PLoS Pathog. 2012, 8, e1003001.
[6] Lee, N.;McGeer, A. Lancet 2020, 395, 1815.
[7]Tay, M. Z.; Poh, C. M.; Renia, L.; MacAry, P. A.; Ng, L. F. P. Nat. Rev. Immunol. 2020, 20, 363.

